Caracterização genética da coleção nuclear do arroz por
marcadores ESTs e SSRs visando a busca por diversidade alélica em genes que
controlam características relacionadas à produtividade e qualidade de grãos.
Vaz, ARC; Brondani, RPV; Rangel, PN;
Borba, T; Grisi, MCM; Louzada, GA; Brondani, C. vaz_arc@yahoo.com.br
Laboratório
de Biotecnologia, Embrapa Arroz e Feijão.
Palavras-chave:
marcadores moleculares, Oryza,
diversidade gênica.
Introdução
A eficiente administração e uso da variabilidade genética contida em coleções de germoplasma de espécies cultivadas é um desafio para todos os programas de melhoramento genético do mundo. A incorporação de um componente genômico funcional no programa de melhoramento genético fornecerá subsídios para se efetuar uma busca direcionada por variabilidade alélica de genes que controlam os caracteres de interesse através da análise em acessos de arroz da coleção de germoplasma (Brondani & Brondani, 2004).
A Coleção Nuclear Brasileira de Arroz (CNBA) é composta por
550 acessos, divididos em 3 estratos: Variedades Tradicionais, genótipos
melhorados brasileiros, e genótipos melhorados introduzidos de outros países. O
estrato mais importante é o de VTs, por representar a varibilidade genética de
um germoplasma adaptado a uma grande diversidade de ambientes onde se cultiva o
arroz no Brasil, e que por isto possuem grande potencial de uso no
melhoramento.
Objetivos
1.
Caracterização
genética de locos ESTs (Expressed Sequence Tags)
e SSRs (Microssatélites) associados a características relacionadas à qualidade
de grãos, visando a identificação da diversidade alélica nos acessos da CNBA;
2.
Gerenciamento da
variabilidade genética existente nos grupos de acessos de arroz de sequeiro e
irrigado para a estruturação de futuros cruzamento utilizando genótipos da CNBA
com maior potencial para transferência dos alelos diferenciais através do
cruzamento com variedades elite de arroz.
Metodologia
Fizeram parte da análise de diversidade genética, 384
acessos de arroz que compõem a CNBA, divididos em 6 grupos: Variedades
tradicionais (VT) de arroz irrigado; VT de arroz de sequeiro; introduções (I)
de arroz irrigado; I de arroz de sequeiro; genótipos melhorados (GM) de arroz
irrigado; GM de arroz de sequeiro.
Para a análise genética foram utilizados marcadores ESTs
relacionados à característica de qualidade de grão, identificados após uma
busca no GenBank; e marcadores SSRs associados a QTLs para produtividade. A
detecção de polimorfismo alélico foi efetuada em géis de poliacrilamida a 4%
corado com nitrato de prata de acordo com Bassam et al. (1991).
Estimou-se o número de alelos
polimórficos e restritos de cada grupo; a heterozigosidade observada (Ho),
diversidade gênica (He) e análise de similaridade genética utilizando os
programas estatísticos GDA, TFPGA e Ntsys (Lewis & Zaykin,
![]()
2000; Miller, 1997; Rohlf, 1989).
Resultados
A análise genética com 11 marcadores ESTs e 9 marcadores SSRs identificou uma média de 4,65 alelos por loco (Figura 1). Os índices médios de He foram sempre maiores para o grupo de acessos de arroz irrigado (44%), e menores para de arroz de sequeiro (23%).
Os acessos introduzidos de arroz irrigado
apresentaram o maior índice de He (50%), enquanto os acessos do material de
melhoramento de sequeiro, os menores (16%). O valor médio encontrado para He
(33%) foi superior ao índice de Ho (6,5%). Ao todo foram identificados 93
alelos.
O dendrograma obtido (Figura 2) utilizando
os dados da matriz de distância
genética (Rogers modficado, r = 0,95) mostra a predominância de dois grandes
grupos, divididos em arroz e sequeiro e irrigado. A distância genética média
entre os acessos de arroz irrigado e de sequeiro foi de 49%, sendo a distância
média entre os acesso de irrigado de 30% e os de sequeiro de 20%.
Polimorfismo de SSRs x ESTs: Marcadores
derivados de locos SSR apresentaram maior informatividade ( He = 0,57) que
locos derivados de sequências expressas - ESTs ( He = 0,40). Apesar de menos
informativos, o uso de marcadores ESTs são importantes por que podem ser
relacionados diretamente com características de importância agronômica, como no
caso da qualidade de grão (frações proteína e carboidrato), priorizada no
desenvolvimento dos ESTs desenvolvidos neste trabalho.
Discussão e Conclusões
Os maiores índices de diversidade gênica
encontrados foram sempre referentes aos acessos de arroz irrigado. Estas
estimativas estão de acordo com o histórico de introdução do arroz no Brasil.
Introduções de arroz irrigado são provenientes de vários locais no mundo e
diversas regiões do continente Asiático, enquanto os acesso de arroz de
sequeiro são oriundo de áreas restritas no continente Asiático;
A grande diferença encontrada na variabilidade
genética destes dois grupos de genótipos melhorados é resultante da utilização
de genitores com maior grau de parentesco no grupo de arroz de sequeiro, uma
vez que o Brasil é um dos únicos países do mundo que possuem um programa para
este ambiente de cultivo, condição totalmente contrária do arroz irrigado;
Existe a necessidade de utilização da
variabilidade genética existente no grupamento VT de arroz de sequeiro e
irrigado brasileiras no programa de melhoramento de sequeiro, por representar
um grupo de acessos geneticamente divergente, e com potencial para ampliar a
base genética das cultivares comerciais de arroz;
Esta forma de selecionar os recursos genéticos
do arroz, priorizando aqueles que possuam alelos diferenciais para genes de
interesse, possibilitará explorar com maior eficiência a variabilidade
genética, culminando com o lançamento de melhores cultivares de arroz.
Acessos de arroz irrigado ainda mantém
variabilidade genética suficiente para viabilizar sua utilização no programa de
melhoramento. Esta informação já está sendo utilizada pelo grupo de melhoristas
para a inserção de novos genótipos no programa de cruzamentos, a fim de
aumentar a probabilidade de ganhos genéticos para características de
importância econômica para o arroz de sequeiro.
Referências bibliográficas
Bassam, B.J.; Caetano-Anolles, G.
and Gresshoff, P.M. (1991) Fast and
sensitive silver staining of DNA in polyacrilamide gels. Analytical Biochemistry 196: 80-83.
Brondani, R.P.V.; Brondani, C. (2004) Germoplasma: Base para
a nova agricultura. Ciência Hoje. V.
35, p 70-73.
Lewis, P.O.; Zaykin,
D. (2000) Genetic Data Analysis: Computer program for the analysis of allelic
data. Version 1.0 (d15) Free program distributed by authors over the Internet
from the GDA Home Page at http://alleyn.eeb.uconn.edu/gda/2000.
Miller, M. (1997) Tools For Population Genetic Analyses
(TFPGA) 1.3: A windows program for analyses of allozyme and molecular
population genetic data.
Rolf, F.J. (1989) NTSYS-Pc: Numerical Taxonomy and
Multivariate Analysis System. Exeter publisher, New York.
Agradecimentos: CNPq/Profix, CNPq/IC e DTI.

Figura 1. Caracterização molecular utilizando marcadores
EST (A) e SSR (B) nos grupos de acessos da CNBA: VT irrigado (1 a 30), VT
sequeiro (31 a 60), GMI irrigado (61 a 69), GMI sequeiro (70 a 78), GMB
irrigado (79 a 87) e GMB sequeiro (88 a 96).
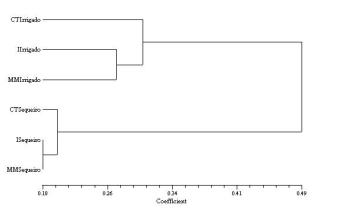
Figura 2.
Dendrograma baseado em freqüências alélicas ilustrando a divergência genética
entre os grupos de acessos avaliados da CNBA.